1. Gefahrenantwort bei polytraumatisierten Patienten
Das Forschungslabor der Klinik und Poliklinik für Orthopädie, Unfallchirurgie und Plastische Chirurgie der Universität Leipzig nimmt an einer nationalen prospektiven Multizentrumstudie des Netzwerkes Traumaforschung (NTF) der Deutschen Gesellschaft für Orthopädie und Unfallchirurgie (DGOU) teil. Von polytraumatisierten Patienten und gesunden Probanden werden Blutproben sowie Daten gesammelt und geteilt. Ziel ist es, potentielle Biomarker zu finden, die den Verlauf, die Prognose und das Outcome von polytraumatisierten Patienten vorhersagen. Im Rahmen dieser Studie werden eigene Forschungsprojekte in Leipzig bearbeitet.
| Studienleitung in Leipzig: | Prof. Dr. rer. nat. Gabriela Aust
Prof. Dr. med. habil. Christian Kleber |
Klinische Mitarbeiter:
(Akquirierung und Aufklärung der Patienten, Blutabnahmen)
| PD Dr. med. Georg Osterhoff
Dr. med. Annette Keß
Carolin Fuchs
|
| Forschungslabormitarbeiter: | Leyu Zheng
Nele Häußler |
1.1 Regulation von PUFAs und deren Metaboliten im posttraumatischen Verlauf
Metabolite von mehrfach ungesättigten Fettsäuren (PUFAs) bilden ein bioaktives Lipid-Netzwerk, das zahlreiche entzündliche and pro-resolving Immunprozesse reguliert. Die Metabolite werden nach Polytrauma aus den geschädigten Geweben und aktivierten (Immun-)Zellen freigesetzt. Dieses Lipid-Netzwerk ist im posttraumatischen Verlauf bisher wenig charakterisiert. Mittels targeted Flüssigkeitschromatographie-Massenspektrometrie (LC-MS) werden PUFA-Metabolite im Plasma von polytraumatisierten Patienten 1 h bis 10 Tage nach dem Trauma quantifiziert und mit den klinischen Daten der Patienten korreliert. Das Projekt wird in enger Zusammenarbeit mit der AG Klinische Massenspektrometrie (Prof. Dr. rer. nat. Uta
Ceglarek, Dr. rer. nat. Madlen Reinicke) des Institutes für Laborationsmedizin, Klinische Chemie und Molekulare Diagnostik (ILM) des Universitätsklinikums Leipzig realisiert.
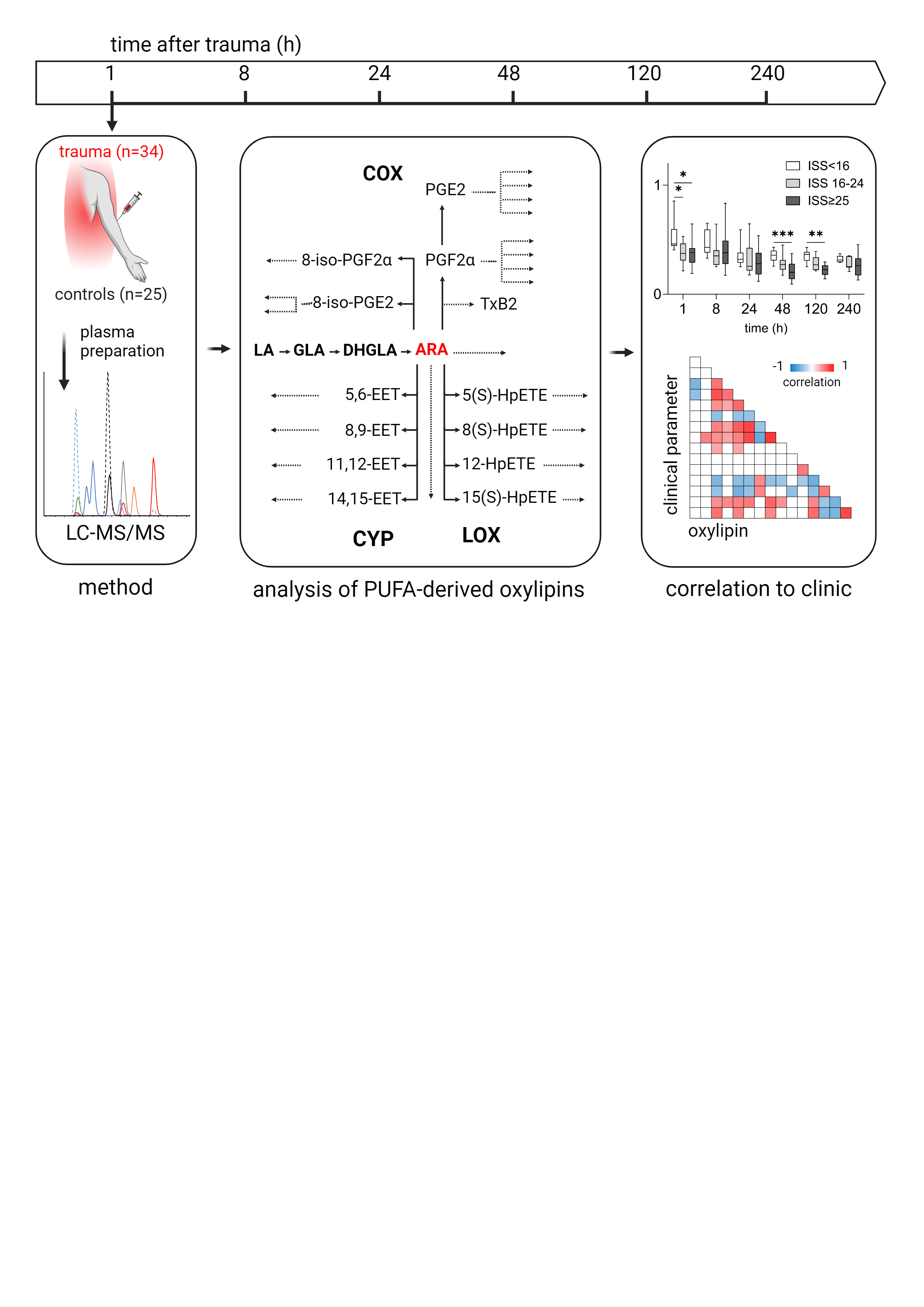 |
| Abb. 1: Ablauf des Projektes "PUFAs und deren Metabolite im posttraumatischen Verlauf" |
1.2 Regulation von Adhäsions-G-Protein-gekoppelten Rezeptoren im posttraumatischen Verlauf
Der klinische Verlauf, die Prognose und das Outcome von polytraumatisierten Patienten wird wesentlich von der (Immun-)Antwort auf die freigesetzten Gefahrsignale und beteiligten Pathogene bestimmt. Adhäsions-G-Protein-gekoppelte Rezeptoren (GPCR) sind Membranrezeptoren mit ungewöhnlich großen extrazellulären Domänen, die u. a. Adhäsion und Migration von Leukozyten vermitteln. Einzelne interessante Vertreter dieser Familie, die auf Subpopulationen der Immunzellen exprimiert werden, sind an der Regulation der angeborenen und adaptiven Immunantwort beteiligt. Die Expression, Aktivierung und Suppression von Adhäsions-GPCR auf Subpopulationen von Leukozyten werden im posttraumatischen Verlauf 1 h bis 10 Tage nach Trauma mittels Durchflusszytometrie analysiert und mit den klinisch-pathologischen Daten der Patienten korreliert. Ziel ist es, Adhäsions-GPCR als potentielle Biomarker des posttraumatischen Verlaufs zu charakterisieren und die Regulation dieser Rezeptoren in Immunzellen nach Polytrauma zu verstehen.
 |
| Abb. 2: Ablauf des Projektes "Regulation von Adhäsions GPCRs im posttraumatischen Verlauf" am Beispiel von EMR2/ADGRE2. |
2. Funktion und Signaltransduktion von Adhäsions-G-Protein-gekoppelten Rezeptoren in der Haut
Adhäsions-G-Protein-gekoppelte Rezeptoren (GPCR) sind eine spannende Familie von Membranrezeptoren, die große extrazelluläre Anteile besitzen. Die verschiedenen Domänen in diesem „Arm" ermöglichen Interaktionen (u. a. Adhäsion = Name) mit anderen Rezeptoren oder mit Bestandteilen der extrazellulären Matrix. Von den 33 humanen Adhäsions-GPCR zeigt nur GPR115 ein selektives Transkriptionsprofil in der Haut. Wir konnten mit einem sorgfältig validierten GPR115 Antikörper zeigen, dass GPR115 in einer kleinen Subpopulation der basalen sowie in allen nicht-verhornten suprabasalen Keratinozyten der normalen Epidermis lokalisiert ist. Deletion von GPR115 in HaCaT Keratinozyten führt zum Verlust des wichtigen Keratins KRT1 und in kultivierten HaCaT-Hautäquivalenten zu einer geringeren Schichtung der Epidermis. Wir charakterisieren z. Z. HaCaT Klone, die eine gain-of-function Mutation in GPR115 aufweisen. Unsere Daten zeigen, dass GPR115 die Differenzierung der Epidermis reguliert.
| Klinische Mitarbeiter: | OA Dr. med. Stefan Glasmacher |
| Forschungslabormitarbeiter: | Nele Häußler
Marianne Quaas
|
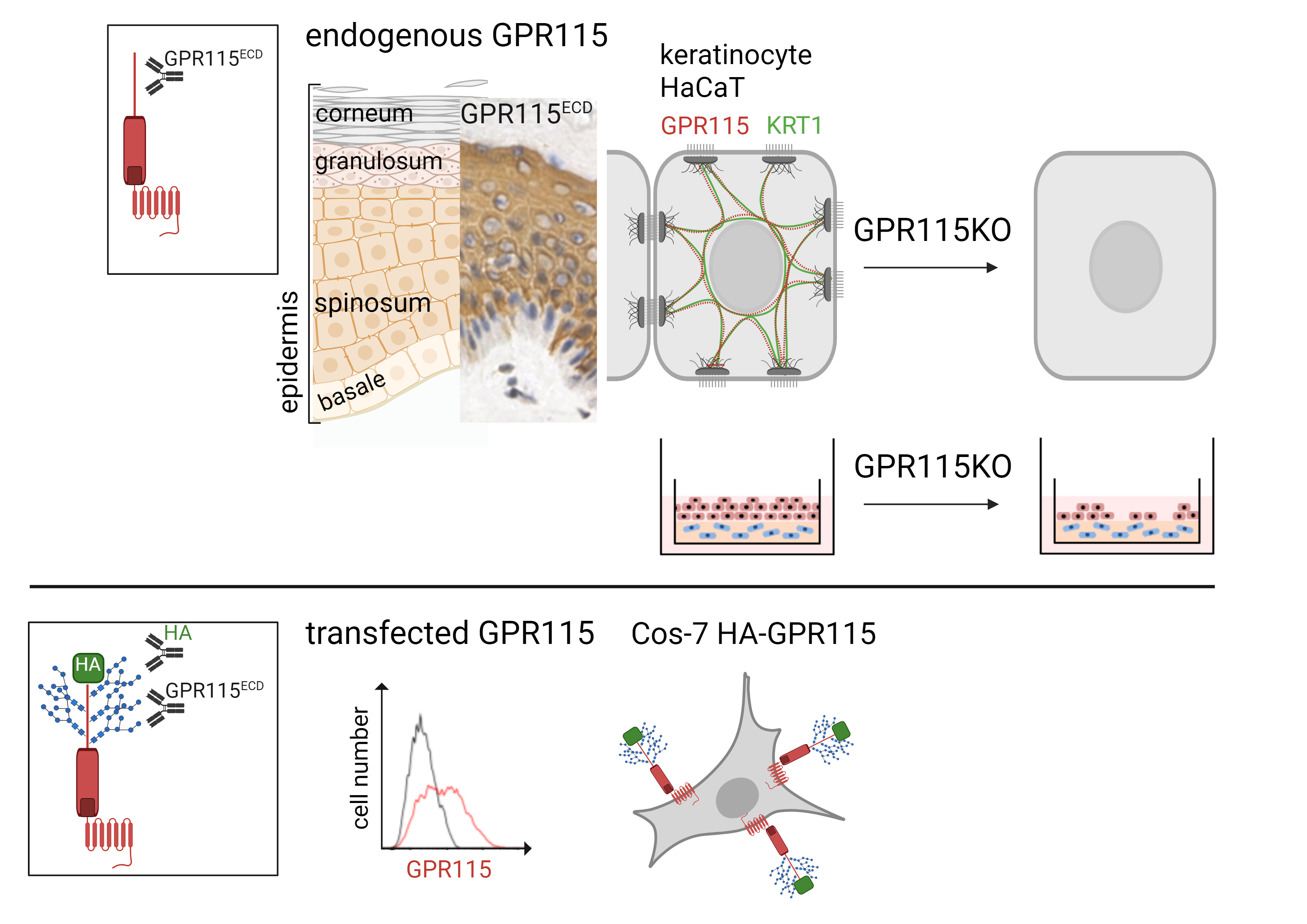 |
| Abb. 3: Projekt "Expression und Funktion des Adhesions GPCRs GPR115 in der humangen Epidermis" GPR115 ist in suprabasalen, nicht-verhornten Keratinozyten vorhanden. Bei Einsatz von GPR115-defizienten HaCaT Keratinozyten ist in vitro die Bildung von Hautäquivalenten gestört. |