NutriScoPe – Automatisierte Diagnostik von Mangelernährung (Nutricional Scoring) bei stationären Patient:innen
Laufzeit: 11/2024 – 10/2026
Finanzierungsart: TMF – Technologie- und Methodenplattform für die vernetzte medizinische
Forschung e.V. & Haushaltsmittel des Universitätsklinikums Leipzig
Projektbeschreibung
NutriScoPe ist eine retrospektive Studie, die untersucht, wie Mangelernährung bei Patient:innen in verschiedenen Klinikabteilungen auftritt und welchen Einfluss eine systematische Ernährungsdiagnostik und -therapie auf den Behandlungserfolg hat.
Mangelernährung betrifft 20-30 % aller stationär aufgenommenen Patient:innen und ist mit längeren Krankenhausaufenthalten, höheren Kosten und erhöhter Sterblichkeit verbunden, besonders bei malignen Erkrankungen und chronischem Organversagen. Um diesen Risiken entgegenzuwirken, wurde am Universitätsklinikum Leipzig (UKL) ein Ernährungsteam eingerichtet, das Patient:innen gezielt während des stationären Aufenthalts betreut.
Um Patient:innen mit erhöhtem Risiko für Mangelernährung zu erfassen, wird am UKL ein standardisiertes Verfahren das Nutritional Risk Screening (NRS) eingesetzt: Patient:innen durchlaufen bei stationärer Aufnahme ein Vorscreening, bei auffälligen Befunden folgt ein Hauptscreening. Bei nachgewiesener Mangelernährung erhalten sie eine gezielte Ernährungstherapie durch das Ernährungsteam. Im Vergleich dazu wird an der Universitätsklinik Jena bislang keine systematische NRS-Erfassung durchgeführt.
Die Studie basiert auf medizinischen Dokumentationsdaten der beiden Kliniken und analysiert die Prävalenz von Mangelernährung, deren Verteilung auf verschiedene Fachabteilungen sowie die Auswirkungen des automatisierten Nutritional Risk Screening auf Therapieerfolg, Krankenhausverweildauer und Komplikationen. Außerdem werden Zusammenhänge mit Alter, Geschlecht und Hauptdiagnosen untersucht, um gezielte Maßnahmen zur Verbesserung der Patientenversorgung ableiten zu können.
Weitere Informationen zum Projekt finden Sie unter: FDPG - Forschungsdatenportal für Gesundheit
MDS-Projektmitglieder
Projektpartner
Ergebnisse
Die Auswertung der Daten zeigte, dass nicht alle Patient:innen mit einem positiven Vorscreening ein anschließendes Hauptscreening am UKL erhielten, was eine Abweichung vom NRS-Protokoll darstellt. In der klinischen Praxis erfolgte das Hauptscreening überwiegend bei Patient:innen mit schwereren Krankheitsverläufen.
Da das Vorscreening auf einer Selbsteinschätzung bei der Aufnahme in die Klinik beruht, war mit Verzerrungen zu rechnen. Auffällige Hauptscreenings wurden insbesondere in der Viszeralchirurgie beobachtet, häufig bei Patient:innen mit malignen Komorbiditäten.
Ein höherer NRS-Score im Hauptscreening korrelierte deutlich mit ungünstigeren klinischen Verläufen. So zeigten sich bei diesen Patient:innen eine erhöhte Mortalität, eine längere stationäre Verweildauer sowie eine höhere Wiederaufnahmerate.
Die folgende Abbildung veranschaulicht die bisherigen zentralen Ergebnisse der Analyse (Stand 09/2025):
Abbildung 1: Anzahl der Patient:innen, die von 2017 bis 2023 ein Nutritional Risk Screening (NRS-Screening) am UKL erhalten haben. Eigene Darstellung.
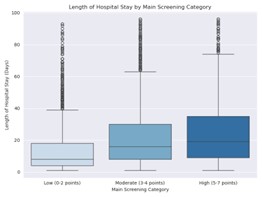 Abbildung 2: Dauer des stationären Aufenthalts in Abhängigkeit von der NRS-Score-Gruppierung im Hauptscreening
| 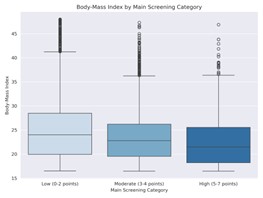 Abbildung 3: Body-Mass-Index in Abhängigkeit von der NRS-Score-Gruppierung im Hauptscreening
|
Publikationen
- Pirlich, M., Schütz, T., Norman, K., Gastell, S., Lübke, H. J., Bischoff, S. C., Bolder, U., Frieling, T., Güldenzoph, H., Hahn, K., Jauch, K. W., Schindler, K., Stein, J., Volkert, D., Weimann, A., Werner, H., Wolf, C., Zürcher, G., Bauer, P., & Lochs, H. (2006). The German hospital malnutrition study. Clinical nutrition (Edinburgh, Scotland), 25(4), 563–572. Read Paper
- Schuetz, P., Fehr, R., Baechli, V., Geiser, M., Deiss, M., Gomes, F., Kutz, A., Tribolet, P., Bregenzer, T., Braun, N., Hoess, C., Pavlicek, V., Schmid, S., Bilz, S., Sigrist, S., Brändle, M., Benz, C., Henzen, C., Mattmann, S., Thomann, R., … Mueller, B. (2019). Individualised nutritional support in medical inpatients at nutritional risk: a randomised clinical trial. Lancet (London, England), 393(10188), 2312–2321. Read Paper